La Biologia Molecolare è una materia molto affascinante, ricca di studi che ci riguardano molto da vicino, trattando dei costituenti del nostro corpo, oltre che ovviamente anche di quelli degli altri esseri viventi. Ma tanto è affascinante quanto intricata, al punto che capire come facciano le cellule a fabbricare le proteine è stata un’impresa talmente difficile che gli studiosi definirono la sintesi proteica “The central dogma of Biology” (il dogma centrale della Biologia). E doveva essere vissuto come un problema davvero complicato se fu usato proprio il termine più odiato in assoluto dagli scienziati: dogma. Cioè verità universale e indiscutibile, cosa a cui si crede ciecamente e che non si pone in discussione. Ma per fortuna questo nome fu assegnato unicamente per esorcizzare la difficoltà e il timore di non riuscire a capire, stante che i dogmi non sono ammessi nella Scienza ed anzi fanno accapponare la pelle di chi vi lavora.
Le proteine sono così importanti, anzi fondamentali, perché sono i principali costituenti che determinano la struttura e la funzione delle nostre cellule. Vengono assemblate proprio all’interno di queste ultime e per capire in che modo si deve partire dal DNA, il famoso acido desossiribonucleico che abita nel nucleo delle cellule (eucariote, alle quali mi riferisco nel resto dell’articolo).
È cosa ben nota che il DNA ha la forma di una doppia elica che si ripiega su se stessa e si inviluppa fino nei cromosomi. Le due eliche presentano dei costituenti, come fossero mattoncini, chiamati ‘nucleotidi’ che si legano a coppie, tenendo in questo modo ben salda tutta la molecola, come visibile in Figura 1:
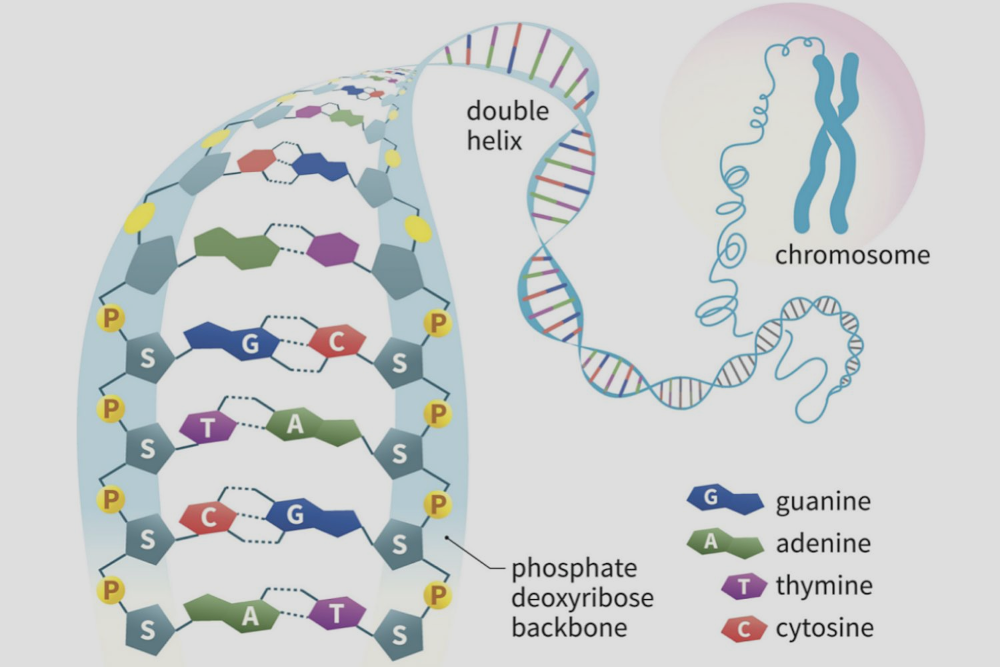
Figura 1. Il DNA in prospettiva, a sinistra uno ‘zoom’ sui suoi costituenti, a destra il multiplo ripiegarsi fin dentro al cromosoma.
Spesso in Natura le cose più belle sono anche le più semplici e il DNA non fa eccezione. Solo quattro infatti sono i tipi di nucleotidi, ognuno dei quali si lega volentieri soltanto ad un preciso omologo:
Guanina (G) con Citosina (C)
Timina (T) con Adenina (A)
Ad esempio, indicando i nucleotidi con le proprie iniziali, se la sequenza di un’elica è GATCACGGA allora quella dell’elica di fronte dovrà essere CTAGTGCCT, il che con le eliche affacciate l’una verso l’altra (come nella realtà) è più facile da capire:
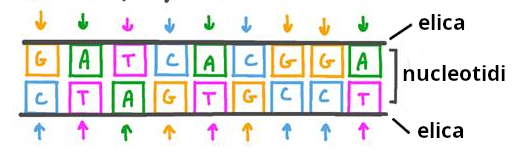
Pur con sole 4 lettere si possono scrivere innumerevoli sequenze tutte diverse le une dalle altre. Ciò significa che il DNA custodisce miriadi di “codici segreti” che le cellule possono decidere di utilizzare al momento opportuno.
Una catena di DNA può essere formata da centinaia di milioni di coppie di nucleotidi e quindi rappresenta un codice lunghissimo in grado di contenere tantissime informazioni. Come confronto, con una ben nota catena formata da 10 simboli (il numero telefonico del nostro cellulare) sappiamo bene quanti numeri diversi uno dall’altro si possano fabbricare variando la sequenza di quei 10 simboli. Bene, il DNA è simile ad un numero telefonico composto da milioni di cifre.
Questo scrigno di codici segreti che le cellule si portano appresso contiene anche la ricetta per fabbricare le proteine: certi spezzoni della doppia elica costituiscono infatti la precisa sequenza di nucleotidi corrispondente al codice intimo di una determinata proteina.
Già capire tutto questo fu un’impresa epica, ma quando gli studiosi ci arrivarono rimase loro da mettere in ordine ancora qualche problemuccio in merito alla sintesi proteica:
1) il DNA sta nel nucleo, ma le Proteine vengono fabbricate al di fuori di esso, sempre dentro i confini della membrana cellulare, ma nel così detto citoplasma. Come fa il codice segretato in un remoto spezzone di DNA a fuoriuscire dal nucleo della cellula ?
2) la molecola del DNA è lunghissima, ma solo corte parti di essa sono la ricetta per fabbricare una proteina. Come vengono individuate la parti ‘buone’ nel bel mezzo di questa lunghissima doppia elica ?
3) quand’anche lo spezzone giusto venga ritrovato, qual è il suo inizio e quale il suo termine ?
Nel caso della sintesi proteica lo scambio dentro-fuori avviene per mezzo di una piccola molecola chiamata RNA Messaggero o mRNA che, partendo dal nucleo supera la membrana nucleare e giunge infine nel citoplasma.
La membrana nucleare contiene infatti piccole zone di passaggio chiamate NPC (Nuclear Pore Complex) che funzionano proprio come guardiani ad un varco, regolando il transito e decidendo chi può entrare nel nucleo e chi ne può uscire. È attraverso di essi che avvengono gli scambi fra il nucleo e il citoplasma, come visibile in Figura 2:
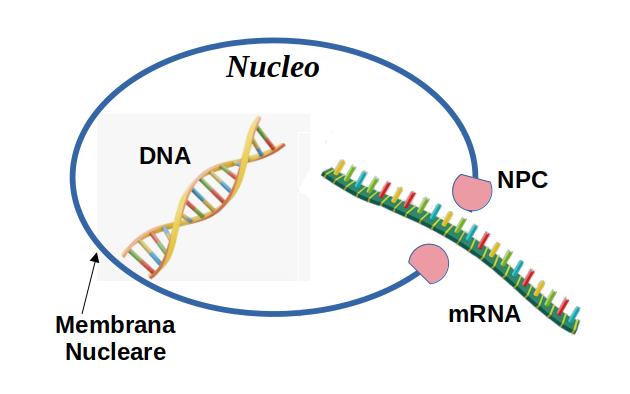
Figura 2. Dai pori presenti sulla membrana del nucleo (NPC) possono transitare molecole. Qui è schematizzata quella dello RNA Messaggero mentre esce dal nucleo.
Prima di uscire, sullo mRNA viene copiato il codice genetico per fabbricare una certa proteina.
È questa la prima delle due fasi importanti del processo, che si chiama trascrizione proprio perché il codice del DNA viene “trascritto” sulla molecola di mRNA. Vediamo come...
La cellula che vuole produrre una nuova proteina attiva una serie di enzimi (ed altri sistemi più complessi) che individuano gli spezzoni della lunga catena del DNA relativi alla proteina da produrre e causano un temporaneo dispiegamento della solitamente compatta doppia elica del DNA. L’attore più importante di questa operazione è un enzima chiamato ‘RNA Polimerasi’ che crea una piccola bolla all’interno del DNA causando il temporaneo allontanamento delle due eliche, un po’ come avviene in una cerniera tipo zip, dove al posto del cursore c’è la ‘bolla di trascrizione’. A differenza della cerniera, la doppia elica a valle rimane chiusa e compatta.
Questo meccanismo è davvero straordinario e fa capire come le cellule siano evolute e in grado di attuare metodi molto sofisticati per produrre tutto ciò che serve loro. A pensarci bene, è come se il prezioso codice genetico venisse gelosamente custodito in uno scrigno impermeabile e protetto (la doppia elica del DNA nel nucleo) e venisse aperto e consultato con saggezza ed estrema cura solo quando necessario. E comunque se ciò avviene, il codice originale si legge e basta, senza perturbarlo minimamente: tutto ciò che è concesso fare è trascriverlo su un foglio (la molecola dello mRNA Messaggero) per richiuderlo nello scrigno subito dopo. Questa visione romanzata farà sorridere un eventuale scienziato o addetto ai lavori che dovesse leggere queste righe, ma è comunque un buon paragone di ciò che avviene realmente.
Il metodo della trascrizione è schematizzato in Figura 3:
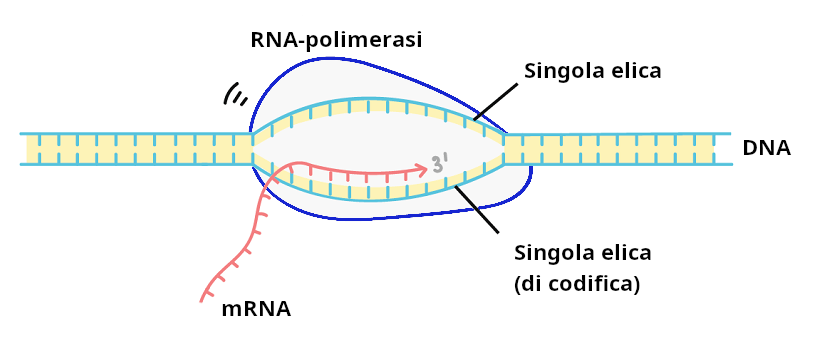
Figura 3. Meccanismo di trascrizione del codice DNA che viene copiato sullo mRNA.
La copia avviene con basi azotate identiche a quelle viste in precedenza per il DNA, con l’unica differenza che la Timina è sostituita da una diversa base che si chiama Uracile. Le coppie sono quindi:
Guanina (G) con Citosina (C)
Uracile (U) con Adenina (A)
Come per il DNA, anche qui diventano dirimpettaie coppie omologhe, ad esempio:
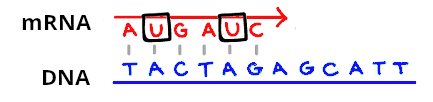
Bene. La ricetta per sintetizzare la proteina è ora disponibile proprio là dove quest’ultima può essere creata, nel citoplasma della cellula, ben trascritta sulla molecola di mRNA che è uscita dal nucleo. E nel citoplasma si trova la vera macchina costruisci-proteine: il ‘Ribosoma’.
Se DNA e RNA sono formati da basi azotate, le proteine sono agglomerati di amminoacidi, che a loro volta sono composti organici. Sebbene ne esistano più di 500, soltanto 22 sono quelli utilizzati nelle proteine e questi 22 amminoacidi corrispondono a specifici codici di basi azotate.
Il Ribosoma legge lo mRNA e lo “traduce” nei corrispondenti amminoacidi: questa è la seconda ed ultima fase che prende infatti il nome di traduzione.
La lettura avviene di tre in tre basi, ovvero in triplette. Ad esempio il Ribosoma può leggere AUC, poi UAA, poi AGG, e così via fino a che l’intera molecola di mRNA è stata percorsa.
A questo punto avviene l’ennesima cosa meravigliosa, entra in gioco l’ultimo attore che possiamo pensare come un facchino che ha sulle spalle un amminoacido e in mano una ben precisa chiave, in grado di infilarsi solo nella propria serratura. Stiamo parlando di un’altra variante di RNA chiamata tRNA o ‘RNA transfer’.
Le molecole di tRNA sono presenti in gran numero nella cellula e vengono richiamate nel Ribosoma quando la tripletta di basi letta è la serratura corrispondente alla loro chiave. Che tradotto in pratica significa: la molecola di tRNA con tripletta di basi azotate esattamente omologa alla tripletta letta in quel momento sulla molecola di mRNA viene richiamata e portata appresso a quest’ultima.
Ricordiamo che “sulle spalle” ogni tRNA ha un ben specifico amminoacido, che a quel punto si trova vicino a quello precedente e che con lui forma dei legami chimici restandovi attaccato. Si forma così, in uscita dal Ribosoma, una catena di amminoacidi che è di fatto la nuova Proteina. La sequenza di amminoacidi di cui è formata, in ultima analisi, era scritta nel DNA !
Il meccanismo è raffigurato in Figura 4:
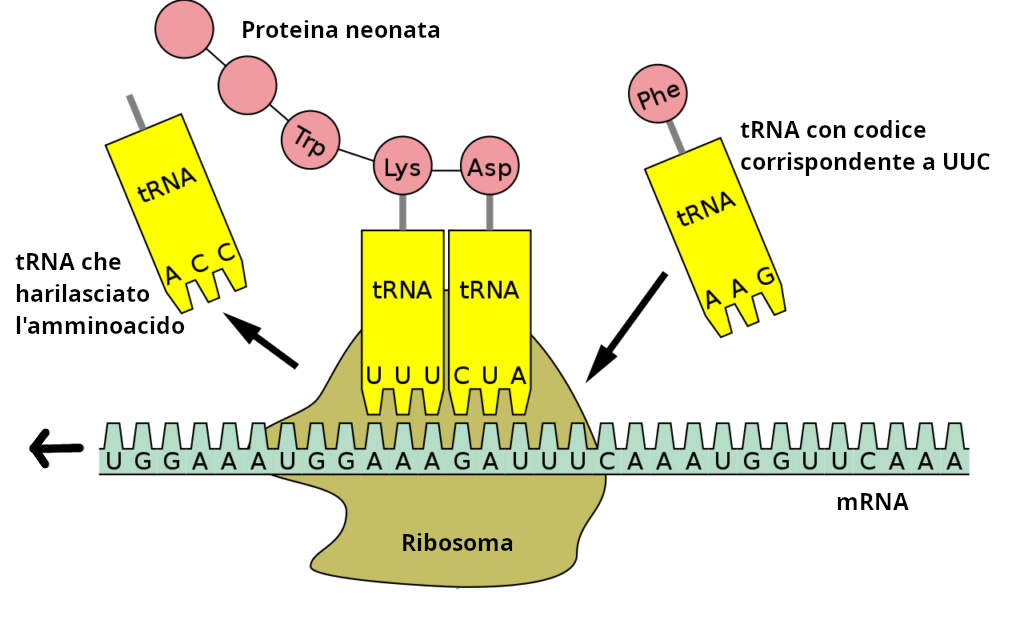
Figura 4. La fabbrica delle proteine. La molecola di mRNA viene letta da destra verso sinistra di 3 in 3 basi. Il Ribosoma richiama la molecola di tRNA corrispondente al prossimo codice di 3 basi, facendo così avvicinare il suo amminoacido al precedente a cui si lega formando la catena proteica.
A questo punto forse l’immagine di copertina, che poteva sembrare la rappresentazione artistica di un qualche mondo extraterrestre, è più chiara e comprensibile. E fa vedere in rosa a destra che la catena polipeptidica fabbricata dal Ribosoma per diventare una vera e propria proteina deve avvoltolarsi più e più volte su se stessa a formare non un disordinato groviglio di amminoacidi, ma una struttura ripiegata ordinatamente in eliche o fogli chiamati rispettivamente alpha-helix e beta-sheets.
Il meccanismo di produzione delle proteine, ingegnoso e perfetto, con cui la Natura è riuscita a stupire l’umanità, dopo essere stato ovviamente motivo di ammirazione si è rivelato anche un’ottima fonte di ispirazione in altri campi del Sapere.
Ad esempio, sebbene i possibili gruppi di 3 basi azotate “pescate” nell’insieme delle 4 possibili (A,U,C,G) siano 4 elevato alla terza potenza cioè 64, ricordiamo che soltanto 22 sono gli amminoacidi utilizzati come costituenti della catena proteica. Questo significa che la cellula ha a disposizione una sovrabbondanza di codici a tre cifre rispetto a quelli strettamente necessari (ne ha 64, ne basterebbero 22). Che farne degli altri ? Ecco alcune arguzie messe in atto.
Innanzi tutto, si sfrutta il concetto di ridondanza per rendere più sicuro o più rapido il processo: molteplici tRNA con codici distinti trasportano il medesimo amminoacido. Ad esempio l’Arginina è codificata indifferentemente dalle triplette CGU, CGC, CGA, CGG. Questo ci ha insegnato che avere a disposizione più codici per una stessa informazione spesso semplifica le cose e le rende più robuste e immuni agli errori di lettura. E così nei protocolli di trasmissione fra computer (o da computer a periferiche) sono state implementate soluzioni molto simili.
Inoltre, esiste un unico codice speciale e ben specifico che non corrisponde ad alcun amminoacido proteico. È il codice AUG, che quando ritrovato sulla molecola di mRNA è utilizzato dal Ribosoma come codice di partenza per la fabbricazione della proteina. Questo metodo è stato decisamente “rubato” al Ribosoma ed implementato nella maggior parte dei protocolli di trasmissione di tipo seriale fra dispositivi elettronici. Questa prevede un unico filo su cui transitano le informazioni digitali in una direzione, ad esempio da un mouse ad un computer e ogni pacchetto di dati viene rilevato grazie al riconoscimento di un “bit di start” che è l’esatto stesso metodo utilizzato dal Ribosoma per dare inizio alla fabbricazione della proteina.
Infine, similmente a quanto avviene all’inizio, esistono particolari sequenze che individuano il codice di termine messaggio. Sono le triplette UAA, UAG, UGA che, come nel caso di quella di inizio, non corrispondono ad alcun amminoacido bensì forniscono al Ribosoma l’informazione che la sequenza è stata completata e che si può terminare il processo di fabbricazione.
In campo informatico questa strategia è stata immediatamente carpita e concretizzata nei “bit di stop” oppure nei “codici di stop”, particolari sequenze che informano il processore ricevente che il messaggio digitale trasmesso è terminato e che ogni bit che dovesse sopraggiungere o è spurio oppure appartiene ad una successiva comunicazione.
Insomma, per concludere, è sempre dalla natura che dobbiamo trarre ispirazione e consiglio. Copiarne metodi e trucchi porta sempre a realizzare cose che funzionano.
Marco Sartore
Nota dell’autore:
casualmente preparai l’articolo sull’Intelligenza Artificiale (vedi Pillole di Scienza 14) proprio nella settimana in cui sull’argomento veniva conferito il Premio Nobel per la Fisica 2024 a John J. Hopfield e Geoffrey E. Hinton che proprio di AI sono stati rispettivamente il pioniere e uno dei massimi studiosi.
Altrettanto casualmente questo articolo è stato preparato nella settimana in cui veniva conferito il Premio Nobel per la Chimica 2024 a David Baker, Demis Hassabis e John M. Jumper, per i loro mirabolanti lavori sulle proteine. In particolare Baker ha dimostrato che è possibile costruire proteine artificiali e i rimanenti due laureati Nobel hanno sviluppato un sistema di AI per la loro analisi.
Come si può intuire quindi, nella Scienza quasi nulla è slegato dal resto, né vive di vita propria. Lavori scientifici di così grande portata, apparentemente lontani fra loro, si coagulano in un ‘unicum’ che rappresenta il varco verso un mondo migliore, in cui ad esempio capiremo come evitare che alcune proteine aiutino il cancro e come altre, a lungo andare, si adattino ai farmaci antitumorali vanificandone l’efficacia.
Riferimenti bibliografici:
Alberts B (2015). Molecular biology of the cell (Sixth ed.). Abingdon, UK: Garland Science, Taylor and Francis Group. ISBN 978-0815344643
Berk A, Lodish H, Darnell JE (2000). Molecular cell biology (4th ed.). New York: W.H. Freeman. ISBN 9780716737063
Dobson CM, The nature and significance of protein folding, in Pain RH (ed.) (a cura di), Mechanisms of Protein Folding, Oxford, Oxfordshire, Oxford University Press, 2000, pp. 1–28, ISBN 0-19-963789-X.
Zhang C, Kim SH, Overview of structural genomics: from structure to function, in Current Opinion in Chemical Biology, vol. 7, n. 1, 2003, pp. 28–32, DOI:10.1016/S1367-5931(02)00015-7